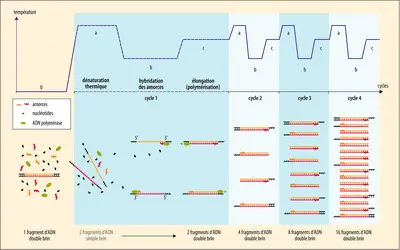
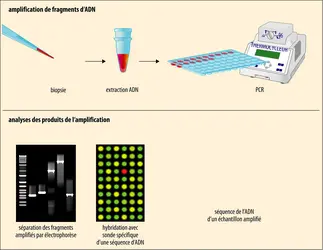
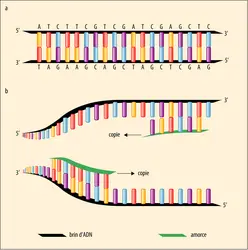
PCR (polymerase chain reaction) ou AMPLIFICATION EN CHAÎNE PAR POLYMÉRASE
La PCR, l'usine flexible des chercheurs
La polyvalence des procédures est le résultat de l'adaptation de la PCR aux questions sans cesse nouvelles des chercheurs. Les applications de la PCR sont de ce fait considérables.
Détermination des séquences d'ADN : l'explosion de la génomique
La plus connue des applications de la PCR est le séquençage des molécules d'ADN, c'est-à-dire la détermination de la succession des nucléotides qui les composent, source d'une quantité considérable d'informations en biologie.
Les techniques de séquençage ont beaucoup évolué depuis les méthodes manuelles des débuts, remplacées par la migration des fragments d'ADN marqués par des sondes fluorescentes à l'intérieur de capillaires dans la première génération d'appareils de séquençage. L'évolution des machines à séquencer a été très rapide et, même si le séquençage direct de l'ADN sans amplification préalable est devenu réalisable, toutes les méthodes exigent de disposer de quantités significatives d'ADN, donc d'ADN amplifié. Pour cela, des techniques de PCR très particulières, adaptées à chaque procédure de séquençage, ont été mises au point.
Dans les technologies de séquençage dites « de deuxième génération », on amplifie la totalité du génome en amplifiant au hasard un très grand nombre de petits fragments qui ne sont pas définis a priori. Ces petits fragments sont ensuite séquencés en parallèle, par centaines de milliers ou de millions. La séquence initiale de l'ADN est reconstituée à partir de ces millions de courtes séquences, grâce à des programmes de la bio-informatique. Toutes les machines actuelles exigent donc une étape d'amplification de l'ADN par PCR rendue indépendante de la séquence génomique, par l'addition aux extrémités des fragments d'une courte séquence d'ADN synthétique qui sert d'amorce universelle à plusieurs centaines de milliers ou de millions de fragments d'ADN. Ces fragments sont isolés physiquement dans des microréacteurs liquides ou sur une surface solide. Cette méthode permet de s'affranchir de la principale limitation de la PCR ciblée : la spécificité des amorces qui nécessite la connaissance a priori de la séquence cible. L'ensemble est rapide et peu onéreux : le séquençage actuel du génome humain coûte environ 500 euros et prend trois jours (entre 2 et 3 milliards de dollars et plusieurs années lors du lancement du programme « génome humain » en 1990).
Tout cela explique l'explosion du nombre de publications de génomes complets d'animaux et de plantes les plus divers. Les banques de données génomiques sont ainsi devenues une mine, tant dans le domaine des biotechnologies que pour les systématiciens et les phylogénéticiens. La comparaison de génomes entiers a ainsi bouleversé les classifications animale et végétale et donné des aperçus tout à fait nouveaux sur les mécanismes génétiques en cause dans les processus évolutifs.
Un bouleversement dans la notion d'identité individuelle et groupale
La recherche en génétique a grandement bénéficié de l'amplification par PCR. Celle-ci permet de se focaliser sur une portion d'intérêt du génome et d'en détecter le polymorphisme (c'est-à-dire toute variation dans la séquence de l'ADN de deux gènes par ailleurs identiques, qu'elle se traduise ou non par un caractère visible) en combinant la PCR à d'autres techniques telles que le séquençage de l'ADN, mais également le repérage d'un polymorphisme déjà connu par hybridation avec une sonde qui en est spécifique. La PCR a ainsi renouvelé la génétique des populations animales et végétales.
Mais la PCR est aussi très largement employée pour réaliser des empreintes génétiques en examinant simultanément plusieurs[...]
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Véronique BARRIEL : maître de conférences au Muséum national d'histoire naturelle, Paris
Classification
Pour citer cet article
Véronique BARRIEL. PCR (polymerase chain reaction) ou AMPLIFICATION EN CHAÎNE PAR POLYMÉRASE [en ligne]. In Encyclopædia Universalis. Disponible sur : (consulté le )
Médias
Autres références
-
POLYMERASE CHAIN REACTION (PCR)
- Écrit par Nicolas CHEVASSUS-au-LOUIS, Universalis
- 296 mots
-
BACTÉRIOLOGIE
- Écrit par Jean-Michel ALONSO, Jacques BEJOT, Michel DESMAZEAUD, Didier LAVERGNE, Daniel MAZIGH
- 18 329 mots
- 11 médias
...une sensibilité permettant de détecter parfois jusqu'à une seule copie d'une séquence génomique spécifique. La méthode la plus connue est la réaction de polymérisation en chaîne, ou P.C.R. (pour polymerase chain reaction). Elle utilise la propriété de l'enzyme ADN polymérase de synthétiser le brin complémentaire... -
BIOLOGIE - La biologie moléculaire
- Écrit par Gabriel GACHELIN
- 7 405 mots
- 8 médias
...définies de plus en plus longues et maintenant d'ARN, ce qui permet nombre de développements techniques. Enfin, une technique majeure introduite en 1985, la polymerase chain reaction (PCR), permet l'amplification d'à peu près n'importe quelle séquence d'ADN à partir de quantités aussi faibles que l'ADN... -
DENGUE
- Écrit par Philippe DESPRÈS
- 2 880 mots
- 4 médias
Pendant la phase aiguë de la maladie, la virémie est recherchée parPCR en temps réel, laquelle utilise des couples d'amorces capables de reconnaître toutes les souches virales ou, au contraire, spécifiques de chacun des sérotypes de la dengue. L'isolement viral à partir d'un sérum virémique est réalisé... -
DIAGNOSTIC VIROLOGIQUE
- Écrit par Yannick SIMONIN
- 5 194 mots
- 4 médias
...et cellulaires, ce qui permet d’extraire le génome viral normalement protégé à l’intérieur des capsides virales ou au sein des cellules infectées. L’amplification génique peut ensuite être effectuée. Elle repose sur latechnique de polymérisation en chaîne (PCR pour polymerase chain reaction). - Afficher les 23 références
Voir aussi
- IDENTITÉ JUDICIAIRE
- ENZYMES
- MICRO-ORGANISME
- HYBRIDATION MOLÉCULAIRE
- HÉRÉDITAIRES MALADIES ou MALADIES GÉNÉTIQUES
- ADN POLYMÉRASE
- ARN MESSAGER ou ARNm
- DIAGNOSTIC PRÉIMPLANTATOIRE
- INFECTION
- SÉQUENÇAGE, génétique moléculaire
- GÉNOME
- BIOLOGIE MOLÉCULAIRE
- AGROALIMENTAIRE TECHNOLOGIE
- DIAGNOSTIC PRÉNATAL
- CYTOLOGIE ou BIOLOGIE CELLULAIRE
- AMPLIFICATION GÉNIQUE IN VITRO
- EXPRESSION GÉNÉTIQUE
- EMPREINTES GÉNÉTIQUES
- NUCLÉOTIDIQUE SÉQUENCE
- RECONNAISSANCE, biologie
- GÉNÉTIQUE MOLÉCULAIRE
- TRANSCRIPTASE INVERSE ou REVERSE TRANSCRIPTASE
- PHYLOGÉNIE MOLÉCULAIRE
- PALÉOGÉNÉTIQUE ET PALÉOGÉNOMIQUE
- ADN ANCIEN
- TAQ POLYMÉRASE