PLASMIDES
Plasmides R : plasmides de résistance aux antibiotiques
Historique
C'est vers la fin des années 1950 que deux microbiologistes japonais, Ochiai et Akiba, observèrent au cours d'une épidémie de dysenterie bacillaire l'apparition de bactéries résistantes à trois antibiotiques à la fois, rendant les malades atteints insensibles au traitement par des antibiotiques habituellement efficaces. Un mécanisme de mutation chromosomique-sélection, auparavant décrit par Ledeberg et qui permettait d'expliquer les résistances bactériennes observées jusque-là, ne permettait pas d'expliquer cette triple résistance simultanée à trois familles d'antibiotiques différentes. Les auteurs japonais montrèrent alors que cette résistance multiple se transférait entre bactéries intestinales dans le tube digestif des malades, puis ils reproduisirent ce transfert in vitro entre colibacilles et shigelles et démontrèrent que ces bactéries résistantes possèdent en plus de leur chromosome d'autres structures composées d'ADN portant les gènes responsables de cette résistance multiple transférable : ce sont les plasmides de résistance aux antibiotiques ou plasmides R. La découverte de ces éléments génétiques extrachromosomiques mobiles apparut d'emblée d'une importance considérable en clinique par leur incidence sur l'arsenal thérapeutique antibiotique. Les plasmides R permettent aux bactéries d'acquérir la résistance à un ou plusieurs antibiotiques d'emblée ; cette résistance est transférable de bactérie à bactérie de la même espèce mais aussi d'espèces voire de genres différents ; la résistance peut apparaître ainsi dans une espèce bactérienne jusque-là sensible, la sélection par un antibiotique peut être responsable de la sélection de la résistance à plusieurs antibiotiques non employés, enfin la coexistence possible de plusieurs plasmides R dans une seule cellule bactérienne permet d'expliquer l'émergence dans les milieux à forte pression de sélection antibiotique comme le milieu hospitalier de bactéries multirésistantes responsables d'échec thérapeutique. Depuis leur découverte chez les entérobactéries, des plasmides R ont pu être mis en évidence chez la plupart des bactéries d'intérêt médical, vétérinaire ou industriel.
Espèces bactériennes hébergeant des plasmides
Depuis leur découverte chez Shigella dysenteriae, des plasmides de résistance ont été isolés dans la plupart des bactéries pathogènes. Des plasmides ont été mis en évidence chez les bacilles à Gram négatif (entérobactéries diverses, Yersinia, Pseudomonas, Acinetobacter, Aeromonas, Vibrio, Haemophilus, Pasteurella, Bordetella, Campylobacter, Bacteroides fragilis), les bacilles à Gram positif (Actinomyces, Bacillus, Clostridium perfringens), les cocci à Gram positif (staphylocoques, streptocoques) et les cocci à Gram négatif (gonocoques). Le pneumocoque et le méningocoque restent les deux seules bactéries de grande importance en pathologie infectieuse dépourvues pour l'instant de plasmides. Des plasmides de résistance peuvent être isolés de nombreuses bactéries de l'environnement, en particulier chez les Streptomyces, bactéries productrices d'antibiotiques. Ces bactéries possèdent les gènes de résistance qui les protègent de l'action de l'antibiotique qu'elles produisent. Ces bactéries productrices d'antibiotiques seraient le réservoir et l'origine de la dissémination des gènes de résistance chez les bactéries pathogènes.
Diversification
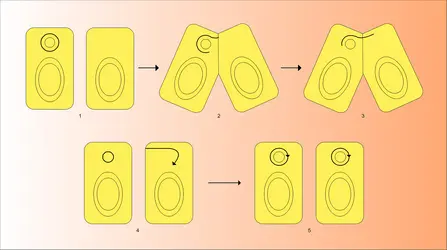
Deux mécanismes, la mutation et la transposition, permettent la diversification du matériel plasmidique.
La suite de cet article est accessible aux abonnés
- Des contenus variés, complets et fiables
- Accessible sur tous les écrans
- Pas de publicité
Déjà abonné ? Se connecter
Écrit par
- Annie BUU HOÏ : docteur en médecine, assistant des hôpitaux, chef de travaux
- Michel GUÉRINEAU : maître de recherche au C.N.R.S.
Classification
Pour citer cet article
Annie BUU HOÏ et Michel GUÉRINEAU. PLASMIDES [en ligne]. In Encyclopædia Universalis. Disponible sur : (consulté le )
Médias
Autres références
-
EUCARYOTES (CHROMOSOME DES)
- Écrit par Denise ZICKLER
- 7 721 mots
- 9 médias
...centrale très riche en adénine et thymine (Fitzgerald-Hayes et al., 1982). Ces fragments d'ADN (de 600 à 800 paires de nucléotides) ont été insérés dans des plasmides, molécule d'ADN circulaire, autoréplicable, qui sont eux-mêmes réintroduits dans une cellule de levure. Grâce à un jeu de marqueurs génétiques... -
ANTIBIORÉSISTANCE
- Écrit par Aurélie CHABAUD, Sylvain MEYER, Marie-Cécile PLOY
- 5 907 mots
- 4 médias
Les plasmides de résistance (en réalité les plasmides porteurs de gènes de résistance) sont les principaux acteurs de la dissémination de ces gènes au sein des populations bactériennes. Les plasmides sont de petites molécules d’ADN circulaire fermées, indépendantes du chromosome bactérien, et capables... -
BACTÉRIES
- Écrit par Jean-Michel ALONSO, Jacques BEJOT, Patrick FORTERRE
- 11 052 mots
- 3 médias
...fonctions non codées par les gènes chromosomiques. Ces ADN extrachromosomiques sont transférables d'une bactérie à l'autre. Il s'agit des plasmides et épisomes. Les plasmides au sens strict sont des ADN extrachromosomiques se répliquant dans le cytoplasme bactérien, indépendamment de la réplication du... -
BACTÉRIOLOGIE
- Écrit par Jean-Michel ALONSO, Jacques BEJOT, Michel DESMAZEAUD, Didier LAVERGNE, Daniel MAZIGH
- 18 329 mots
- 11 médias
...génétique consiste à l'amplification des gènes en amenant les structures qui les portent à être répliquées rapidement. On multiplie ainsi les plasmides, petits morceaux d'ADN circulaires extrachromosomiques au nombre de 2 à 30 par bactérie. Dans certaines espèces, pour amplifier des gènes et... - Afficher les 13 références
Voir aussi
- HYBRIDATION MOLÉCULAIRE
- RÉPLICATION, biologie moléculaire
- SHIGELLA
- EUCARYOTES
- ALIMENTATION ANIMALE, élevage
- AZOTE ATMOSPHÉRIQUE
- BIOLOGIE MOLÉCULAIRE
- RHIZOBIUM
- AZOTE CYCLE DE L'
- CHROMOSOMES
- PROCARYOTES ou PROTOCARYOTES
- RÉSISTANCE BACTÉRIENNE
- STREPTOMYCES
- ÉPISOMES
- ÉPIDÉMIES
- ENZYMES DE RESTRICTION ou ENDONUCLÉASES DE RESTRICTION
- CONJUGAISON BACTÉRIENNE
- GÉNÉTIQUE MOLÉCULAIRE
- TRANSPOSON